Importance
La détection des interactions moléculaires est à la base de nombreuses applications biotechnologiques importantes dans la société et l'industrie, telles que la découverte de médicaments, les diagnostics et le séquençage de l'ADN. Ce rapport décrit une plate-forme largement applicable pour détecter les interactions moléculaires à l'échelle d'une seule molécule, en temps réel, sans étiquette et de manière potentiellement hautement multiplexable, à l'aide de capteurs à une seule molécule sur une puce de réseau de capteurs à semi-conducteur hautement évolutive. Ces puces sont à la fois pratiquement manufacturables à court terme et ont une feuille de route de mise à l'échelle durable à long terme, offrant ainsi un moyen idéal d'apporter la puissance de la technologie des puces modernes au vaste domaine de la biodétection. Ce travail concrétise également une vision scientifique vieille de 50 ans consistant à intégrer des molécules uniques dans des puces électroniques pour parvenir à la miniaturisation ultime de l'électronique.
Abstrait
Depuis près de 50 ans, la vision de l'utilisation de molécules uniques dans les circuits a été considérée comme la miniaturisation ultime des puces électroniques. Un exemple avancé d'une telle puce électronique moléculaire est présenté ici, avec la distinction importante que les éléments de circuit moléculaire jouent le rôle de capteurs à molécule unique à usage général. Le dispositif se compose d'une puce semi-conductrice avec une architecture de réseau évolutive. Chaque élément de matrice contient un fil moléculaire synthétique assemblé pour couvrir des nanoélectrodes dans un circuit de surveillance de courant. Un site de conjugaison central est utilisé pour attacher une seule molécule sonde qui définit la cible du capteur. La puce numérise la lecture résultante du courant en fonction du temps à l'échelle du picoampère de chaque élément de capteur du réseau à une fréquence de 1 000 images par seconde. Cela fournit des signatures électriques détaillées des interactions d'une seule molécule entre la sonde et les cibles présentes dans un échantillon de test en phase solution. Cette plate-forme est utilisée pour mesurer la cinétique d'interaction de molécules uniques, sans l'utilisation de marqueurs, de manière massivement parallèle. Pour démontrer une large applicabilité, des exemples sont présentés pour la liaison de la molécule sonde, y compris les oligos d'ADN, les aptamères, les anticorps et les antigènes, et l'activité des enzymes pertinentes pour le diagnostic et le séquençage, y compris une enzyme CRISPR/Cas se liant à un ADN cible et une ADN polymérase enzyme incorporant des nucléotides lorsqu'elle copie une matrice d'ADN. Toutes ces applications sont réalisées avec une sensibilité et une résolution élevées, sur une puce semi-conductrice entièrement électronique, évolutive et manufacturable, apportant ainsi la puissance des puces modernes à ces divers domaines de la biodétection.
Des mesures rapides, spécifiques et sensibles des analytes cibles sont les objectifs de nombreuses méthodes utilisées en biologie moléculaire et en biotechnologie. Les méthodes en vrac utilisent généralement une molécule de liaison pour reconnaître la molécule cible, combinée à des mécanismes rapporteurs optiques indirects, tels que des marqueurs de colorant fluorescent ou des modifications des propriétés optiques en vrac résultant de la liaison à la cible. De telles méthodes classiques détectent une moyenne sur de nombreux événements de liaison moléculaire et sur des échelles de temps beaucoup plus longues que celles des interactions moléculaires primaires. En revanche, les interactions de liaison d'intérêt se produisent fondamentalement au niveau de la molécule unique et sont généralement dynamiques et stochastiques dans le temps, et contiennent donc beaucoup plus de détails que ce qui se reflète dans les taux de réaction en vrac. Cela met en évidence la possibilité d'accéder à un niveau d'information fondamentalement plus riche et plus puissant lors de la mesure des interactions moléculaires.
Les approches d'observation des détails des interactions d'une seule molécule entrent dans des catégories basées sur la méthode de détection (1). Beaucoup sont des biocapteurs optiques à molécule unique basés sur la fluorescence (2⇓⇓–5), bien que d'autres techniques physiques spécialisées aient été utilisées, notamment des capteurs électrochimiques (6, 7), des capteurs plasmoniques (8), la spectroscopie Raman à surface améliorée (9) , et des méthodes couplées à la détection des nanopores (3, 10, 11). En plus des complications des procédures d'étiquetage nécessaires pour ajouter des reporters fluorescents aux cibles d'intérêt, les méthodes optiques à molécule unique souffrent de limitations fondamentales en matière de signal et de résolution. Un défi majeur pour les méthodes de fluorescence à molécule unique est d'obtenir un rapport signal sur bruit élevé, car le taux de production de photons à partir de molécules de colorant unique est limité par les limites d'intensité d'éclairage et le photo-blanchiment (12), contraignant à la fois à court et à long terme. -mesures de temps. Les effets de mouvement moléculaire et la diffraction limitent également la résolution spatiale ultime ou la densité des rapporteurs optiques multiplex.
Passer de la détection basée sur les photons à la détection entièrement électronique peut supprimer ces contraintes fondamentales sur le rapport signal sur bruit, la mise à l'échelle et la bande passante, et est de plus compatible au maximum avec la mise en œuvre sur les puces semi-conductrices modernes. Il serait avantageux de mesurer les interactions moléculaires sur une puce semi-conductrice métal-oxyde complémentaire (CMOS) pour tirer parti de leur fabrication de masse à faible coût, de leur vitesse et de leur miniaturisation. Ce sont les caractéristiques des appareils modernes à base de puces CMOS, tels que les ordinateurs portables et les téléphones portables. Ces dispositifs sur puce bénéficient également d'une feuille de route durable pour les améliorations futures fournies par 50 ans de mise à l'échelle des puces CMOS selon la loi de Moore, ainsi que de l'infrastructure de fonderie de puces et des chaînes d'approvisionnement correspondantes.
Cependant, le plein potentiel de cette vision de la biodétection moléculaire mobile "sur puce" ne peut être réalisé qu'en utilisant un concept de capteur compatible de manière appropriée. À cette fin, trois principes fondamentaux de conception de capteurs doivent être pris en compte : la fabricabilité, l'évolutivité et l'universalité (voir l'annexe SI). Le domaine de l'électronique moléculaire fournit une solution conceptuelle à tous ces défis, dans laquelle une seule molécule dans un circuit fournirait le capteur entièrement mis à l'échelle pour résoudre le problème de mise à l'échelle de More-than-Moore commun aux dispositifs de détection. Les progrès scientifiques sur les propriétés électriques des molécules, ainsi que les inspirations bioélectroniques, ont conduit à la proposition au début des années 1970 selon laquelle des molécules uniques pourraient être conçues pour être utilisées comme éléments de circuit (13), pour exécuter des fonctions de circuit telles qu'un redresseur ou un interrupteur. En raison des limites de la technologie de nanofabrication, ce n'est qu'à la fin des années 1990 que les premiers circuits à une seule molécule ont été démontrés expérimentalement (14). L'intérêt pour ce domaine de l'électronique moléculaire s'est considérablement développé après ce point (15⇓⇓–18), et il a été proclamé la percée scientifique de l'année par Science en 2000 (19). Là, il a été noté que l'intégration de molécules dans les puces serait l'avancée critique nécessaire pour que ce nouveau domaine ait un large impact.
L'étude expérimentale de la détection électronique à une seule molécule était initialement basée sur des dispositifs de détection à nanotubes de carbone (NTC). Leur potentiel en tant que capteurs d'interactions à une seule molécule est apparu (20) initialement dans le contexte de la détection de molécules de gaz (21) puis de réactions chimiques (22). Nuckolls, Shepard et ses collègues (23⇓–25) ont introduit un capteur à molécule unique pour les processus de liaison ADN-ADN (hybridation), en fonctionnalisant un CNT avec une seule molécule «sonde» d'oligomère d'ADN. Collins, Weiss et ses collègues (26⇓–28) ont montré qu'un CNT peut être utilisé pour surveiller en temps réel l'activité d'une seule molécule enzymatique attachée au nanotube, y compris les enzymes ADN polymérase. Malheureusement, à l'heure actuelle, il n'existe aucun moyen de fabriquer en masse des NTC ayant une structure et des fonctionnalisations précises, et malgré des décennies d'attention (20), il n'y a pas non plus de voie établie pour les intégrer dans des dispositifs à puce CMOS manufacturables (29, 30). Ainsi, alors que les capteurs à fil moléculaire CNT ont permis des travaux pionniers sur la détection d'une seule molécule, ils ne satisfont pas aux principes de conception d'une plate-forme de capteur à puce CMOS.
En revanche, un fil moléculaire idéal devrait permettre à l'ingénierie de précision de fournir une fraction de conjugaison spécifique au site pour la fixation des molécules de sonde, ainsi que de fournir des groupes terminaux appropriés pour l'auto-assemblage dans les nanoélectrodes sur une puce CMOS, et devrait être facilement disponible. grâce aux procédés de fabrication existants. Cela limite les candidats aux peptides, protéines ou ADN en tant que fils moléculaires, car ce sont en fait les seuls polymères conducteurs pour lesquels il existe des capacités de synthèse de précision bien développées, y compris des moyens étendus de fonctionnalisation de précision. Les hélices d'ADN double brin (dsDNA) (31⇓⇓⇓–35) et les hélices α de protéines (36⇓⇓⇓⇓⇓⇓⇓⇓–45) ont toutes deux été étudiées en tant que fils moléculaires. Des exemples de mesures de courant continu à travers diverses hélices α (courtes) dans la littérature (42) suggèrent qu'une hélice α (longue) de 25 nm pourrait présenter des courants dans la large gamme de 3 picoampères (pA) à 120 pA à une polarisation de 1 volt , en fonction de la séquence d'acides aminés, des conditions de tampon et de la nature de l'attachement peptide-métal. Des méthodes détaillées de sonde à effet tunnel ont également été récemment utilisées pour étudier la conduction à travers des protéines plus grosses (46⇓⇓–49). Bien qu'ils ne soient pas aussi conducteurs que les NTC, ces biopolymères ont le grand avantage pour les besoins actuels de permettre une ingénierie de précision utilisant la capacité de fabrication existante.
Résultats
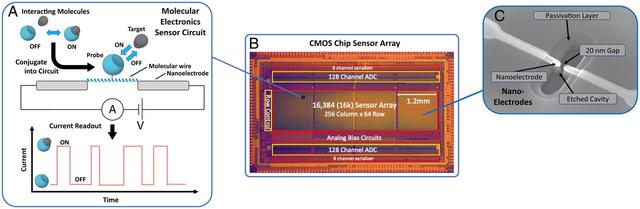
La puce présentée ici est une puce de circuit intégré CMOS qui prend en charge un réseau évolutif de capteurs électroniques moléculaires à base de nanoélectrodes, comme illustré sur les Fig. 1 et Annexe SI, Fig. S1. Ces capteurs sont basés sur un «pont» de fils moléculaires couvrant un espace d'environ 20 nm entre les pointes des nanoélectrodes, qui se connectent elles-mêmes à un circuit de surveillance du courant pour une mesure en temps réel du courant par rapport au temps traversant le pont. Pour une application de capteur spécifique, la molécule pont est conjuguée de manière site-spécifique avec une biomolécule d'intérêt, qui agit comme une sonde pour ses interactions moléculaires avec une molécule cible. Les impulsions de courant observées représentent les changements de résistance électrique induits par ces interactions dynamiques. Étant donné que ces capteurs sont des molécules uniques isolées, ils révèlent les événements de liaison de manière discrète et avec une sensibilité élevée, sans avoir besoin de marqueurs. Le résultat est une mesure électrique directe des interactions moléculaires avec la cinétique correspondante.
Fig. 1.Concept de puce et de capteur électronique moléculaire. (A) Concept Senor : étant donné une paire de molécules qui subissent une interaction, l'une des paires est sélectionnée comme molécule sonde et conjuguée à un fil moléculaire de précision (ici une protéine α-hélicoïdale synthétique) qui couvre un espace nanométrique entre les nanoélectrodes métalliques . Ceux-ci le connectent à une source de tension d'entraînement et à un circuit de surveillance du courant pour fournir une lecture en temps réel du courant par rapport au temps, pour le courant traversant le complexe fil moléculaire/sonde. Lorsque la molécule cible se lie à la sonde, la résistance du complexe change, entraînant un changement observé de courant. La trace de courant résultante a des impulsions marche/arrêt qui fournissent une représentation directe des interactions moléculaires. (B) Dispositif à puce CMOS : un réseau de capteurs à grande échelle est fabriqué à la surface d'une puce CMOS. Montré est une image annotée du dispositif à puce CMOS utilisé dans ces études. Cette puce possède 16 000 capteurs et les circuits nécessaires pour numériser et transférer les lectures des capteurs hors puce, à une vitesse de 1 000 images par seconde. (C) Image SEM de nanoélectrodes de capteur, montrant l'écart de 20 nm pour le pont moléculaire. Les nanoélectrodes présentées sont fabriquées par photolithographie, en utilisant un procédé compatible avec la fonderie CMOS.
Fig. 2.Le capteur de liaison d'hybridation d'ADN. (A) Une sonde oligo d'ADN simple brin (ssDNA), ici l'oligonucléotide 17-mer (5′-TACGTGCAGGTGACAGG-3′), est conjuguée au pont en utilisant la chimie de clic conventionnelle à l'extrémité 5′. (B) Exemple de trace de courant en fonction du temps, montrant 6 s de données échantillonnées à 1 kHz, prises avec le capteur exposé à une concentration de 20 nanomolaires (nM) de l'oligo cible, ici le brin 14-mer complémentaire (5′-CCTGTCACCTGCAC ), en suspension dans une solution tampon standard. Chaque impulsion de courant au-dessus de la ligne de base représente un seul événement de liaison à l'ADN. Les durées des événements et le temps entre les événements sont stochastiques, avec des distributions exponentielles telles que résumées dans l'annexe SI, Figs. S2–S4. (C) La distribution des valeurs de courant mesurées dans la trace, affichée à côté de la trace sous forme d'histogramme vertical, fournit une visualisation du temps passé lié (courant plus élevé) et non lié (courant inférieur). Dans cet exemple, la fraction du temps passé lié est de 22 %. (D) Réponse du capteur de liaison d'ADN à la concentration cible : liaison de la sonde d'ADNsb 17-mer sur le capteur à la cible 14-mer, à des concentrations cibles de 10 nM, 100 nM et 1 000 nM. La largeur des pics (temps de séjour) reste constante (~ 25 ms), mais le temps entre les pics diminue de 45 à 4 ms à mesure que la concentration augmente, reflétant des interactions plus fréquentes axées sur la concentration. La fraction de temps dans l'état lié (marquée « fraction liée » dans les tracés) a été estimée à partir des histogrammes des valeurs de mesure actuelles (encarts à droite). (E) Mesures du temps de séjour et de la fraction liée par rapport à la concentration. Comme prévu pour la liaison par hybridation d'ADN, le temps de séjour reste constant avec la concentration, mais la fraction de temps liée montre une courbe de saturation classique, à partir de laquelle est calculée une affinité de liaison, Kd , de 39 nM. (F) Courbes de fusion thermique à une seule molécule dérivées du capteur de liaison à l'ADN. Dans cette expérience, une sonde d'ADNss 45-mer (5′-CGATCAGGCCTTCACAGAGGAAGTATCCTGTCGTTTAGCATACCC-3′) est attachée au pont à l'extrémité 5′. Deux oligos cibles complémentaires différents, dont les propriétés correspondent étroitement mais qui ont des points de fusion différents, ont été construits en utilisant une cible 15-mère (5′-CCTCTGTGAAGGCCT) du 45-mère, et une extension de celle-ci à une cible 20-mère (5 ′-CCTCTGTGAAGGCCTGATCG). Ceux-ci ont été ajoutés à la puce à une concentration de 20 nM, réalisés en série. Pour chaque solution, la température de la solution a été balayée, par paliers de 2 °C, de 41 °C à 55 °C. Des courbes de fusion d'ADN standard ont été ajustées et utilisées pour dériver les points de fusion empiriques, Tmobs , indiqués . Les résultats sont en accord avec les prédictions de volume classiques pour la différence de T m entre les oligos, mais ici sont mesurés entièrement dans un contexte de molécule unique. (G) Spécificité du capteur de liaison à l'ADN pour l'ADN incompatible. Le capteur a été utilisé pour sonder des cibles incompatibles, en utilisant la sonde d'hybridation 45-mer. Les cibles étaient des 20-mères ayant 0, 1, 2 ou 3 mésappariements comme indiqué (séquence en médaillon), ajoutés séquentiellement. Le résultat montre une tendance à la baisse significative de la fraction liée, à mesure que le nombre de nucléotides mésappariés augmente.
Chaque circuit de capteur (ou "pixel") est un ampèremètre dédié, implémenté comme un amplificateur de transimpédance CMOS qui amplifie les courants à l'échelle des picoampères (pA) en tensions à l'échelle du millivolt (mV). Le gain de l'amplificateur est de 5 GΩ, de sorte que chaque picoamp de courant à travers la molécule est amplifié à 5 mV de tension de sortie. Le circuit amplificateur mesure en effet le courant traversant la biomolécule. La plage dynamique de mesure est de 0 à 400 pA, et l'amplificateur est conçu pour être hautement linéaire sur cette plage, avec un bruit subpicoamp (Annexe SI, Fig. S1 A et B), afin de préserver les formes détaillées des impulsions du capteur pour analyse ultérieure. Les circuits de capteurs sont disposés sur la puce avec un pas de 20 μm, pour un réseau total de 16 384 (16k) capteurs dans une zone de seulement 1,2 mm sur 4,8 mm (Fig. 1). La puce transfère les courants analogiques mesurés du réseau de capteurs vers des convertisseurs analogique-numérique sur puce pour un transfert numérique hors puce, à une vitesse de 1 000 images par seconde et avec 11 bits de précision numérique pour chaque lecture de courant. La puce actuelle est fabriquée dans une fonderie CMOS utilisant un processus de fabrication de «nœud» de 180 nm (le nœud est vaguement lié à la taille minimale des caractéristiques du transistor sur la puce, et plus directement lié à la densité relative des circuits). Comme le montre l'annexe SI, Fig. S1 A – D, les nanoélectrodes sont fabriquées dans une deuxième phase de traitement, à l'aide de processus et d'outils de photolithographie compatibles avec la fonderie CMOS, pour permettre une intégration complète de la fabrication dans les fonderies de puces CMOS commerciales existantes. De plus amples détails sur la puce sont fournis dans (Annexe SI, Fig. S1 C–F).
Pour illustrer l'étendue des différents types d'interactions moléculaires pouvant être détectées sur la plate-forme, ce rapport présente des sondes qui détectent la liaison de l'ADN (Fig. 2A), ainsi que des molécules de protéines, de petites molécules, des aptamères et des anticorps (Fig. 3 A, C, E et G), et des sondes pour surveiller l'activité des enzymes qui sont importantes pour les applications et la biologie fondamentale, par exemple les enzymes CRISPR/Cas (Fig. 3I) et l'ADN polymérase (Annexe SI, Fig. S6A). L'annexe SI, Fig. S7 A – F montre des modèles de ruban détaillés de toutes ces constructions de capteurs. L'imagerie directe de plusieurs molécules de pont est également illustrée dans l'annexe SI, Fig. S7G.
Fig. 3.Une enquête sur divers capteurs électroniques moléculaires pour la liaison et l'activité enzymatique, montrée à l'échelle dans les rendus moléculaires, ainsi que les résultats expérimentaux récapitulatifs correspondants. (A–D) Cinétique de liaison des protéines et des petites molécules. En tant que système modèle pour montrer la liaison aux protéines et la liaison aux petites molécules, ce capteur est configuré pour observer (A) une ADN polymérase se liant à une amorce/matrice, et (C) un nucléotide se liant dans la poche de la polymérase. Pour ce système modèle, une matrice d'ADNss 17-mer est conjuguée au pont peptidique à son extrémité 5 '(et avec l'extrémité 3' bloquée pour empêcher la polymérase de se lier à ce site). Un brin d'amorce 14-mère complémentaire est ensuite lié à celui-ci, à l'extrémité distale du 17-mère, pour créer un site d'amorce sur le capteur avec le 3'-OH disponible pour la liaison à la polymérase. (B) Résumé cinétique (temps de séjour, fraction de temps lié) pour la liaison de l'ADN polymérase de Klenow au site de l'amorce, car la concentration de polymérase est titrée de 0,008 à 3,8 µM, dans un fond d'amorce 14-mer de 100 nM pour supprimer la dissociation de l'amorce. L'affinité de liaison déduite de la polymérase est Kd = 530 nM. (D) Un titrage des nucléotides est effectué pour observer la liaison dans la poche polymérase, dans un tampon non catalytique afin d'observer la cinétique de liaison sans incorporation. Une matrice 45-mères se trouve sur le pont et une amorce 31-mères était liée à l'extrémité distale de la matrice, de sorte que la première base de la matrice (A) est complémentaire du nucléotide testé (T). Le nucléotide a été ajouté à des concentrations de 2,5, 5 et 15 µM avec la polymérase 100 nM et l'amorce en présence d'un tampon contenant 10 mM de Sr 2+ (sans Mg 2+ ), dans lequel l'incorporation de nucléotide ne peut pas se produire. Dans ce tampon, le dNTP se liera et se dissociera à plusieurs reprises de la poche de polymérase, et la cinétique de liaison récapitulative résultante est indiquée. Cela sert également à illustrer la détection de la liaison de petites molécules. (E et F) Capteurs d'aptamères : Des capteurs d'aptamères ont été construits, ciblant ici la protéine S du SRAS-CoV-2 avec un aptamère d'ADN de 57 mères (E) et ciblant la protéine N du SRAS-CoV-2 avec un aptamère d'ADN de 97 mères (Annexe SI, Fig. S7B-2), toutes deux issues de la littérature. (F) Les courbes de titrage de réponse à la concentration pour les capteurs aptamère S et N aptamère, pour une gamme de concentrations de protéines cibles appliquées. Les affinités de liaison, Kd , dérivées de ces courbes (6,4 nM, 39 nM) sont similaires à celles rapportées pour les essais de liaison d'aptamère en vrac standard en solution. (G et H) Capteurs anticorps-antigène. En tant que système modèle, une paire antigène-anticorps fluorescéine-antifluorescéine a été utilisée, l'antigène fluorescéine étant présenté sur le capteur en l'attachant au pont à l'aide d'un oligo ADNsb comme lieur. Un oligonucléotide 45-mer a été utilisé, avec l'extrémité 3' (distale) de l'ADN coiffée d'une fluorescéine lors de la synthèse. Un anticorps commercial antifluorescéine (Fab) a été ajouté dans du tampon TKS sur la puce. Les cinétiques récapitulatives sont présentées pour le temps de séjour et la fraction de temps lié, car la concentration d'anticorps est titrée sur la plage indiquée. L'affinité de liaison déduite était Kd = 1,3 uM. Il a été observé que tous les signaux de liaison ont été éteints lorsque 4 mM de fluorescéine libre ont été ajoutés pour saturer l'anticorps, vérifiant la spécificité du signal de liaison. (I et J) Un capteur d'activité enzymatique CRISPR/Cas. Pour assembler une enzyme Cas en tant que sonde sur le pont, un ARN guide ciblant une cible d'ADNdb pour une enzyme CRISPR/Cas12a a d'abord été conjugué au pont, et ceux-ci ont été assemblés sur une puce. Une enzyme Cas12a a été fournie en solution et a permis de s'ancrer aux ARN guides sur les ponts, la programmant ainsi pour l'ADNdb cible, et également de l'attacher efficacement au pont en tant que sonde. Pour ces expériences, l'ARN guide est un 40-mer, attaché au peptide pont en utilisant la chimie du clic au 13e nucléotide, qui est la base qui s'étend le plus à l'extérieur de l'enzyme dans la boucle pseudoknot. La cinétique est résumée dans la courbe de titrage, montrant la fraction de temps liée à la saturation lorsque la cible d'ADNdb varie en concentration, en présence d'une concentration de 20 nM d'enzyme Cas12a libre (non ciblée). Ainsi, cette configuration agit directement comme un capteur pour la cible d'ADNdb, sans évaluer l'activité de la nucléase simple brin non spécifique posttarget-binding. Cette dernière activité non spécifique est également observable sur le capteur lorsqu'il est pourvu d'un substrat ssDNA. L'affinité de liaison observée pour la cible d'ADNdb est Kd = 3 pM. Le tampon expérimental était Tris.HCl 20 mM pH 8,0, KCl 20 mM, SrCl2 10 mM, DTT 4 mM. Deux exemples de traces de signal brut pour D (petite molécule) et E (protéine) sont inclus dans l'annexe SI, Fig. S7 B et F, montrant que le caractère des signaux de courant de pont est similaire pour ces diverses sondes.
Tous ces capteurs ont un format commun, constitué d'une molécule sonde précisément conjuguée à un fil moléculaire (Fig. 2A). Le pont de fil moléculaire utilisé dans ce travail est un peptide α-hélicoïdal, de 25 nm de longueur, avec un site de conjugaison spécifique conçu dans la chaîne latérale d'un acide aminé près du milieu du peptide pour la fixation des molécules sondes, et spécifique au métal des groupes de conjugaison (1) conçus sur les extrémités pour une liaison sélective et un auto-assemblage aux nanoélectrodes métalliques. Lorsque la molécule de pont est livrée aux nanoélectrodes par diffusion passive, un tel assemblage peut prendre environ 24 h (par exemple, en utilisant une concentration de pont de 20 nM et un tampon dilué [2 mM Tris pH 7,5]). En revanche, pour tous les travaux présentés ici, un protocole de diélectrophorèse « à pontage actif » est utilisé pour accélérer et améliorer considérablement le processus d'assemblage. La diélectrophorèse est un processus très efficace dans lequel les forces électriques attirent les molécules polarisables vers l'espace entre les micro- ou nanoélectrodes (31, 35, 36, 50, 51).
Dans le présent travail, cela permet d'effectuer l'assemblage en moins de 10 s. Le protocole de piégeage diélectrophorétique repose sur l'application d'une tension alternative, sous la forme d'une onde sinusoïdale de 100 kHz, 1,6 V crête à crête, appliquée pendant 10 s, suivie d'une mesure de courant continu pour évaluer la formation de ponts. Jusqu'à 10 cycles de ce type sont appliqués dans le processus de pontage. Ces tensions de piégeage sont appliquées simultanément à toutes les nanoélectrodes sur la puce, en commutant des circuits de commande CA sur puce. Comparé à la diffusion passive, le piégeage diélectrophorétique raccourcit le temps de pontage à ~ 10 s (~ 10 000 fois plus rapide), tout en travaillant simultanément à des concentrations d'entrée ~ 1 000 fois plus faibles de molécules de pont. Cette amélioration de la vitesse de réaction suggère que le champ de piégeage augmente efficacement la concentration locale des ponts à proximité des espaces entre les électrodes d'au moins 1 million de fois. Pour évaluer l'efficacité du pontage, le courant continu du capteur après le pontage est comparé à la valeur précédente, et un saut de courant suffisant indique un pontage réussi. Une population de capteurs montrant une augmentation substantielle du courant de pont est ainsi observée, typiquement plus de 10 % de tous les pixels disponibles sur la puce, indiquant la présence du pont peptidique de 25 nm couvrant l'espace entre les électrodes.
Liaison d'hybridation oligo-ADN et validation de capteur.
Les performances du capteur et de la puce sont validées ici en utilisant le système modèle bien étudié d'un oligo d'ADN simple brin comme sonde pour l'hybridation à son brin complémentaire. Il s'agit d'un système de référence utile, car la liaison oligo a été largement étudiée, à la fois empiriquement et théoriquement, y compris au niveau de la molécule unique à l'aide de capteurs CNT (23⇓–25).
Pour la sonde, un oligonucléotide d'ADN 17-mère a été attaché de manière spécifique au site de précision à l'acide aminé central sur le pont (Fig. 2A), en utilisant la chimie classique du clic (Matériels et méthodes). Une fois que les molécules de pont (avec sonde) sont attachées aux électrodes de la puce, le courant de base est mesuré, qui est généralement de plusieurs picoampères sous des tensions appliquées dans la plage de 700 à 1 000 mV. Tout en continuant à surveiller les courants du capteur, l'oligonucléotide 14-mère complémentaire "cible" est ajouté à une concentration particulière. Comme le montre la figure 2B, le capteur sur puce répond à la présence de la cible en solution avec des impulsions de courant qui peuvent être interprétées comme des événements de liaison individuels entre la sonde conjuguée ssDNA et son brin cible complémentaire présent en solution. Ceux-ci existent dans un équilibre dynamique entre les états liés et non liés. Des expériences de contrôle révèlent que cette liaison change avec la concentration cible (Fig. 2D) et la température (Fig. 2F), comme on pourrait s'y attendre pour l'hybridation ADN-ADN.
Analyse des données de liaison d'une seule molécule à l'aide de modèles de Markov cachés.
Les modèles de Markov cachés (HMM) sont utilisés pour quantifier les traces de signal primaires. Les HMM ont déjà été appliqués avec succès à des données de séries chronologiques d'expériences de biophysique à molécule unique (52⇓⇓⇓–56). Dans le cas présent, le HMM attribue les états liés et non liés "cachés" du capteur à des segments de la trace de signal observée qui ont des niveaux de courant statistiquement différents, généralement avec des courants plus faibles pour l'état non lié et des courants plus élevés pour l'état lié. (Voir l'annexe SI, Matériels et méthodes pour les détails du HMM et l'estimation des paramètres cinétiques . ) annexe, figures S2 et S3). Ces temps obéissent à des distributions exponentielles (Annexe SI, Figs. S2 et S4), comme prévu pour une cinétique de réaction chimique de premier ordre à deux états. Une statistique récapitulative pratique est la fraction totale du temps passé dans l'état lié (notée « fraction liée » dans toutes les figures ; par exemple, Fig. 3 B, D, F, H et J). Ceci est vu à l'échelle avec la concentration selon une courbe de saturation classique de Michaelis – Menten (Matériels et méthodes). L'ajustement de cette courbe permet le calcul de l'affinité de liaison classique de l'interaction, Kd , qui, au niveau de la molécule unique, peut être définie comme la concentration cible à laquelle la molécule sonde unique passe un temps égal lié et non lié. La fraction de temps passée dans les états liés et non liés peut également être facilement visualisée à l'aide d'histogrammes verticaux de toutes les valeurs de courant mesurées dans un segment de trace de signal, comme indiqué à droite des traces sur les Fig. 2 C et D.
Thermodynamique monomoléculaire : courbes de fusion.
Une autre application du capteur consiste à déterminer la température de fusion, T m , du duplex d'ADN, qui est définie au niveau de la molécule unique comme la température à laquelle la molécule d'ADN sonde passe des quantités égales de temps dans les états liés et non liés, pour une concentration donnée d'oligo cible. Comme le montre la figure 2F, la mesure de la fraction de temps liée à huit températures permet d'adapter les données à une courbe de fusion d'hybridation d'ADN classique. Pour ces expériences, une plaque chauffante Peltier en contact thermique direct avec la puce est utilisée pour régler successivement différentes températures, les températures mesurées pour la puce étant fournies par des capteurs de température embarqués. D'après ces courbes, il est clair qu'un oligo cible de 20-mères fond à une température plus élevée qu'un sous-segment de 15-mères, comme prévu. La détermination de T m sert à valider que le capteur mesure la réaction de liaison d'hybridation ADN-ADN comme prévu, et a également une valeur pratique dans la sélection d'une température de fonctionnement appropriée pour effectuer des mesures de nombreux événements de liaison. Comme cela se fait classiquement, cette courbe de fusion d'une seule molécule peut être utilisée comme modalité de mesure pour améliorer la spécificité de détection pour la cible d'intérêt, ou pour caractériser les cibles qui contiennent des mésappariements.
Sensibilité à l'inadéquation.
La trace du signal de la sonde de liaison à une seule molécule contient des informations riches sur la réaction de liaison et est également très sensible à la cible de liaison spécifique. Ceci peut être illustré en détail pour la liaison oligo-ADN en examinant l'impact des mésappariements à une seule base introduits dans la séquence oligo cible. Comme le montre la figure 2G, quatre variants d'un ADN cible 20-mère ont été fabriqués, ayant de 0 à 3 bases mésappariées. Avec chaque mésappariement supplémentaire, la fraction liée diminue (et le taux de décalage k off augmente). Ces différences de cinétique de liaison pourraient être encore amplifiées en effectuant les mesures à une température plus proche de la T m de la cible adaptée, ou en effectuant une courbe de fusion de température, comme sur la figure 2F. Cette sensibilité aux mésappariements peut avoir des applications pour les tests de liaison à l'ADN dans lesquels des variants de séquence par rapport à une sonde de séquence de référence pourraient présenter un intérêt, comme la détection de nouvelles souches d'un génome viral, la détection de mutations somatiques dans un génome cancéreux ou la détection de variants de génotype SNP. .
Mécanisme de génération de signal de capteur.
Des expériences de biophysique CNT avec une sonde ADN (23) ainsi que des sondes enzymatiques (26, 57) montrent que le mécanisme de génération de signal dominant dans les capteurs CNT à paroi unique est un effet de champ, où les champs électriques émanant du complexe cible-sonde modifient la circulation du courant dans le fil CNT de manière action à distance. Ceci est démontré en augmentant la concentration de sel pour filtrer les champs électriques dans la solution, et en montrant que le signal est réduit à partir de groupes de charge plus éloignés que la longueur de blindage (Debye) (23). Pour le présent capteur électronique moléculaire, des expériences similaires ont été menées pour examiner l'effet de l'augmentation de la concentration en sel dans le tampon de liaison sur la force du signal et le temps de séjour, la concentration en sel (KCl) augmentant de 2 mM à 2 000 mM (la longueur de Debye diminuant de 6,8 nm à 0,22 nm). Comme le montre l'annexe SI, Fig. S5, les intensités du signal (définies ici comme des hauteurs d'impulsion) diminuent de près de 70 % avec la concentration en sel. Ceci suggère que la part prédominante des modulations de courant observées est due à l'effet de champ mis en évidence pour les capteurs NTC. Il peut y avoir d'autres formes mineures de modulation de conduction également impliquées. Par exemple, ceux-ci peuvent concerner des changements directs dans les voies de conduction électronique à travers le complexe moléculaire, ou des changements dans les nuages d'ions dans la solution autour des molécules, ou des changements d'état électrochimique sur le capteur moléculaire, tels que des réactions de transfert de charge redox. In any case, one benefit is that the sensor allows for measurements of binding even in biologically relevant high salt conditions, much higher than is possible for CNT sensors (23).
Protein and Small-Molecule Binding.
Fig. 3 A and C show two binding processes fundamentally related to DNA polymerase: the binding of the protein to a 3′-OH primer site (Fig. 3 A and B), and the nucleotide substrate binding in the active pocket of the enzyme (Fig. 3 C and D). These particular sensor modalities are useful for the study of polymerases, but they also serve to illustrate detection of protein binding (here polymerase docking to a priming site) and small-molecule binding (here a dNTP interacting with the polymerase binding pocket). The latter could be considered as a model for a small-molecule drug interacting with a binding pocket on a protein target. To construct this sensor, a DNA template oligo is first tethered to the bridge, as above in the DNA binding studies. A complementary primer oligo is then bound to this, to form a primer site, which can then act as a probe for binding a polymerase. Titration of Klenow DNA polymerase binding to the primer complex on the sensor produces a typical saturation curve (Fig. 3B), and a resulting binding constant, K d . The binding of a nucleotide substrate to the binary complex of primer-polymerase is measured (Fig. 3D) in a noncatalytic buffer (lacking Mg 2+ ), so that the polymerase cannot incorporate the nucleotide. Under these conditions, the sensor measures the dynamic equilibrium of the nucleotide substrate entering and exiting the binding pocket, producing a corresponding binding signal trace, and titration saturation curve (Fig. 3D).
Aptamer–Target Binding.
A DNA aptamer is an oligomer with a sequence empirically selected to bind a specific protein or molecule of interest. Such aptamers can be attached to the sensor bridge as probes in exactly the same manner as the oligonucleotides used for DNA binding probes (Fig. 3E). The use of aptamer binding probes provides the capability to detect a great diversity of possible targets. In particular, aptamer technology is well-suited for rapidly developing binding probes for protein targets, for use in targeted protein detection and identification, and proteome characterization and profiling.
One particularly timely application of such protein-binding aptamers is in testing for infectious disease pathogens (58). In particular, the COVID-19 pandemic has highlighted the need for cost-effective, highly specific, rapid, and distributed testing for viral particles. Aptamers against the SARS-CoV-2 N gene protein (nucleocapsid phosphoprotein) (59) and the S gene protein (surface glycoprotein or Spike protein) (60) have been described in the literature, which exhibit the selective and high-affinity binding necessary for a diagnostic test. Antigen tests have been developed using these aptamers, which show high accuracy in detecting SARS-CoV-2 infection (61). To test the use of aptamers on the present sensor chip platform, both the N and S protein aptamers were attached to the bridge (Fig. 3E and SI Appendix, Fig. S7 B and B-2) to study their binding kinetics against SARS-CoV-2 proteins. Using aptamer-functionalized bridges on chips, the corresponding N or S protein target concentration was titrated in solution (Fig. 3F). The measured binding kinetics parameters from chip experiments for the N protein were k off = 40 s −1 , k on = 6.2 × 10 8 M −1 s −1 , and K d = 64 nM. Similarly, measured parameters for the S protein were k off = 35 s −1 , k on = 3.3 × 10 9 M −1 s −1 , and K d = 6.1 nM. These data are in good agreement with the reported affinities for the N protein and S protein aptamers, which were 20–200 nM and 5.8 nM, respectively.
Antibody–Antigen Binding.
To demonstrate antibody–antigen binding, one option is to place the antigen on the bridge and observe the binding to the cognate antibody present in solution. A model system for this is to use a fluorescein dye molecule as the antigen, and a commercially available antifluorescein antibody, as illustrated in Fig. 3G. The fluorescein is conveniently mounted on the bridge using a DNA oligo as a linker, attached by the same chemistry as described above, and where the DNA oligonucleotide is synthesized to have the fluorescein molecule at its 3′ (distal) end to present the antigen. A commercially available antifluorescein antibody was titrated on this chip and the binding activity is summarized in Fig. 3H, resulting in an apparent K d of 1.3 µM. A DNA oligo tether, such as used here, is one convenient means of conjugating antigen molecules to the bridge; however, many well-known methods of conjugation can be incorporated into the bridge molecule design, to support attaching antibodies or antigens, in particular conjugation methods that are compatible with existing antigen or antibody libraries.
CRISPR/Cas Enzyme Activity.
The CRISPR/Cas enzymes originally used for gene editing (62, 63) have recently been proposed as tools for sensitive DNA detection for diagnostics and other applications (64⇓⇓–67). In general, a CRISPR/Cas enzyme binds to a short-guide RNA strand that serves to program it for sequence-specific binding and activation, for example, to cleave the specified target DNA strand. The ability to monitor the single-molecule kinetics of these enzymes could be useful to understanding their multiple complex activities, and also may help in enzyme evolution studies to provide high-throughput screening for useful mutant phenotypes (68⇓–70). In addition, such enzymes have potential uses for diagnostics, based on monitoring for indications that the programmed enzyme has bound its specific target. The originally discovered Cas9 enzyme has been widely adopted for gene-editing functions, but more recently discovered Cas enzyme families—such as Cas12, Cas13, and Cas14—have been proposed for diagnostics (64, 66, 67), since they undergo more dramatic and readily detectible transformations after encountering their target, and therefore simplify the proposed optical reporter methods. However, the single-molecule sensor presented here is capable of directly observing the primary DNA target capture, and thus any of these enzymes could potentially be used diagnostically, and in a highly multiplex target fashion, on these sensor array chips.
Shown in Fig. 3 I and J are results from binding experiments using the CRISPR/Cas12a enzyme (64, 71), which is commonly used as the basis for such diagnostics, programmed by a guide RNA designed to detect a 20-base DNA sequence taken from the S gene of the SARS-CoV-2 virus. The resulting guide RNA bridges were assembled onto the chip, which was then first used to observe titration of the Cas12a enzyme binding to the guide RNA, over a protein concentration range of 0 to 1 µM. Binding was observed when the guide RNA was attached to the bridge at its 13th nucleotide (which corresponds to the outermost exposed point in the pseudoknot loop) (SI Appendix, Fig. S7D) but was not observed when the attachment was at the 3′ terminus of the guide RNA. It is likely that this latter configuration results in steric hinderance to Cas binding, based on the known Cas12a-guide RNA structure (Fig. 3I and SI Appendix, Fig. S7D). The chip with such guide RNA bridges was incubated with 20 nM Cas12a to bind and program the enzyme, and then titrated with 0.1 to 64 pM concentrations of target dsDNA, done under non-catalytic buffer conditions in order to observe the target binding kinetics. A binding affinity of 3 pM was observed for dsDNA binding to the Cas12-guide RNA complex, similar to the results reported for such enzymes (64, 65, 71, 72). Extending the concept illustrated here, tethering distinct guide RNAs to the bridges of distinct sensors on the array provides a means to deploy multiplex targeted CRISPR/Cas enzymes on chip, and monitor their primary detection activity as single molecules, in parallel. This capability could provide for highly multiplexed CRISPR diagnostics, or for massively parallel phenotype screening for Cas enzyme evolution (68).
DNA Polymerase Enzyme Incorporation Activity.
In this case, a single Phi29 DNA polymerase (73) molecule is conjugated to the molecular bridge, using a site-specific conjugation method. The resulting sensor-chip device is provided with a primed DNA template and the required dNTPs for the polymerase to extend the primer on the template. This sensor configuration, illustrated in Fig. 4 and SI Appendix, Fig. S7E allows single-molecule observation of the polymerase activity as it binds and incorporates nucleotides in real time. Exemplary signal results are shown in Fig. 4 and in more detail in SI Appendix, Fig. S6, which show a 25 s portion of the sensor signal trace having ~40 pulses with dwell times longer than ~10 ms. Inspection of this signal trace suggests that each major pulse represents a nucleotide binding and incorporation event, consistent with the known behavior of DNA polymerase and with CNT observations of single-molecule polymerase activity (26⇓–28). For the DNA template chosen here, primer extension should result in 15 dCMP incorporations followed by 25 dAMP incorporations. Visual inspection(Fig. 4) suggests there are two distinctive segments of pulses distinguished by features such as the pulse height, pulse width, and waiting time between pulses. These data segments are therefore putative A-pulse series and C-pulse series, which highlight the potential to distinguish A and C incorporation events. For a more detailed quantitative analysis showing 95% distinguishability of these pulses, see SI Appendix, Fig. S6A and related Methods.
Fig. 4.DNA Polymerase Activity Sensor. A phi29 DNA polymerase is conjugated to the sensor bridge using the SpyTag-SpyCatcher conjugation scheme shown. The 25-second-long signal trace shows an isolated burst of sensor activity that occurs after adding a primed 40-mer template (sequence 5'-25T-15G-3') and corresponding (dCTP and dATP) nucleotides. The expectation is the polymerase would acquire a template and incorporate 15 C's followed by 25 A's. A series of ~40 discrete major pulses are seen, representing putative incorporation events. The signal trace has ~15 wide-spaced, narrower pulses on the left (green region), and ~25 closely spaced, broader pulses on the right (red region), suggesting these are the C and A events, respectively, and that therefore the C and A incorporations events can be distinguished by examining pulse features.
Shown in SI Appendix, Fig. S6 B and C (and SI Appendix, Table S3) are more extensive pulse discrimination analyses, wherein a total of seven feature metrics of each pulse are automatically extracted, and subsequently assessed in a principal component analysis (PCA). When the extracted pulses from the trace are displayed using the two dominant PCs (SI Appendix, Fig. S6B), they fall into two nearly disjoint clusters based on these shape features. These clusters also correspond to the pulses from the putative oligo-dC segment (SI Appendix, Fig. S6B, coded green) and oligo-dA segment (SI Appendix, Fig. S6B, coded red) of the signal trace. Thus, the pulses are largely distinct in their features, consistent with the hypothesis that dAMP and dCMP incorporations produce distinctive pulses. The PCA importance weighting chart (SI Appendix, Fig. S6C) shows the relative contribution of the various shape features to the first PC (SI Appendix, Table S3). This example illustrates the potential for sequencing DNA using such a DNA polymerase activity sensor, based on analyzing the detailed single-molecule kinetics and signal pulse features of the polymerase as it copies a template.
Sensing in Complex Backgrounds: A Model Viral Detection Assay.
The basic detection of molecular interactions demonstrated above can be used to develop many applications. One such example is viral detection, which has been highly relevant to the COVID-19 pandemic. However, for use in practical diagnostic tests, it is important that a sensor be able to reject complex backgrounds of off-target molecules. Ideally, the sensor would be able function in crude samples, such as saliva. To test this, we constructed a model assay mirroring the Center for Disease Control and Prevention's (CDC) issued recommendation for a qPCR test for the COVID-19 infection, based on testing for the presence of two sequences in the N gene (74). PCR products were made using the CDC forward and reverse primers for SAR-CoV-2, with a synthetic plasmid for the N gene serving as the positive control target template, which was spiked into contrived samples. The sensor chip was prepared with a ssDNA oligo probe targeting one strand of the PCR product. This resulting PCR product, unpurified, was applied to the chip to assess the ability of the DNA hybridization sensor to reject complex backgrounds and still detect its specific target, as well as to work with the complex mixtures produced by PCR, as would arise in practical diagnostic tests (Fig. 5). The oligo probe on the bridge is a 21-mer (CCGCATTACGTTTGGTGGACC) taken from the CDC qPCR TaqMan probe sequence (2019-nCoV_N1-P: FAM-ACC CCG CAT TAC GTT TGG TGG ACC-BHQ1) (74). Various target sequences tested against this probe are shown in SI Appendix, Table S2.
Fig. 5.Using a DNA binding sensor to detect a viral target under mock assay conditions. The DNA binding probe chip is used to detect a target DNA PCR product produced from a contrived sample, with confounding complex background. (A) The titration curves show chip sensor response, for various concentrations of targets, for targets consisting of a synthetic 24-mer positive control, an unpurified PCR product from a contrived saliva sample, and this PCR product with salmon sperm DNA added at 2 μg/mL, to mimic the impact of having background genomic DNA contamination in saliva. This high, complex background had little impact on the results. (B) A 3 s signal trace at the lowest concentration tested, 100 pM, showing 5.2% fraction of time bound. This illustrates the strong signal spikes, and the potential to detect much lower concentrations through longer observations.
Each of these targets (SI Appendix, Table S2) were tested for binding on chip and found to conform to positive and negative controls, as expected. Only the last example (double stranded PCR product) displayed weaker binding than the others, suggesting it has partially renatured (thereby lowering the concentration of target single strands) prior to testing on chip.
Targets were tested at concentrations of 10 pM, 100 pM, 1 nM, and 10 nM in buffer A. The PCR products registered similar sensor responses to pure oligo target samples (Fig. 5A), even though they represent a much less pure sample because of PCR off-target byproducts and reagents. In order to further assess the impact of complex background materials, buffer A was mixed with heat-inactivated human saliva (from 10% by volume to 50% by volume), and in another trial it was mixed with a high concentration of highly complex background DNA: salmon sperm DNA at a concentration typical for DNA contamination of saliva (2 µg/mL) (Fig. 5A). Neither one of these challenges had substantial impact on the sensor readout, showing that the sensor is highly specific for its target and robust against complex and even crude saliva samples.
In terms of the overall sensitivity, inspection of the signal at the lowest tested concentration, 100 pM, shows (Fig. 5B) the signal consists of clear pulses of ~6 pA magnitude occurring approximately eight times per second, but bound just 5% of the total time. This target concentration corresponds to ~1 femtomole of oligonucleotide applied to the chip. Based on the trace shown, this is not the lower limit of on-chip detection: at even lower concentrations, by observing such a sensor for longer time, or observing multiple such sensors in parallel for the same target, it would be possible to observe a statistically meaningful number of binding event pulses at much lower target concentrations. This provides a powerful means to reduce the limits of detection for such a test, potentially by up to six orders of magnitude.
Discussion
The molecular electronics sensor chip presented here has a number of powerful features, as well as broad potential for future applications. In particular, as a sensor platform, it has a unique combination of broad applicability, scalability, and single-molecule sensitivity, while the CMOS chip format also provides for manufacturable realization of sensitive, multiplex, rapid, low-cost tests, on compact instruments. These combined features could enable attractive near-term applications, from drug discovery to diagnostics to DNA sequencing. Moreover, it may provide these with a long-term, faster-than-Moore's Law scaling path to ever lower costs and greater speeds for highly durable technology solutions. These points are briefly discussed further in the following sections.
Sensitivity.
The molecular electronics sensor has intrinsic single-molecule detection sensitivity. This is a relatively unique capability in biosensing, where methods that have single-molecule detection capability often rely on biological signal amplification (such as in PCR or ELISA) to increase the signal to the point where it can be readily detected. Having the platform based on a true single-molecule sensor provides the potential for the ultimate limits of sensitive detection, with or possibly without combination with biological signal amplification for various assays. Moreover, while not tested here directly, it may be possible to further increase sensitivity by electronic target amplification using dielectric forces to concentrate targets, as was used here to increase bridge-electrode assembly efficiencies as much as a million-fold.
It is known that such highly sensitive electronic amplifier systems can experience random telegraph noise (RTN) resulting from, for example, single trapped charges changing state. Indeed, the signal pulse trains produced by the molecular electronics sensor (eg, Fig. 2B) look similar to RTN. Such spurious RTN noise modes do indeed appear sporadically on the present type of sensors, and therefore it is important that experiments be designed with controls to distinguish these spurious noise modes from proper detection signals. One useful method is to titrate the concentration of the target and verify that the sensor pixel responds properly to this titration. For example, Fig. 2D shows the output for different target concentrations for the DNA hybridization sensor. This can thereby rule out pixels subject to RTN that could otherwise confound measurements. In addition, the dwell times for RTN pulses will not match the expected dwell time or temperature response for the target of interest, and this can further be used to reject RTN artifacts. Such calibrations and quality controls should ideally be built into assay protocols.
Specificity.
The molecular electronic sensors can provide highly specific detection, as long as the primary molecular interaction is specific. This is perhaps surprising, given the extreme sensitivity of the sensors. This specificity is best demonstrated with the DNA hybridization binding sensors, where the strength of the DNA binding reaction is in effect sequence-programmable, and finely tuned off-target interactions can be studied by introducing mismatched bases in the target. The sensors were challenged with such complex interactions to demonstrate the limits of specificity. As shown in Fig. 2G, a matching oligonucleotide target produces a signal trace distinguishable from one possessing even a single mismatching nucleotide. The difference in affinity is readily apparent by observing the fraction of time bound and also dwell time. This also has implications for practical assays, since such binding probes can therefore be used to discern the presence of a SNP for genotyping, or to detect isolated mutations in viral genomes for viral strain detection, or for cancer genome mutation analysis. At the other extreme of specificity, the sensor demonstrated the ability of the probe to reject highly complex off-target backgrounds, such as genomic DNA and crude saliva. This has implications for practical assays, enabling them to be robust and have simple, rapid sample preparation.
Multiplex Measurements.
The generality of the sensor platform was demonstrated by measuring enzyme activity (DNA polymerase and Cas12a nuclease), and binding of aptamers (for SARS-CoV-2 S and N proteins), an antigen (fluorescein) with antibody target, as well as binding of proteins and small molecules. The scalable pixel array chip therefore brings powerful and practically unlimited multiplexing capabilities to all of these types of detection. For example, it was shown that a DNA binding probe could detect a gene from SARS-CoV-2; through such multiplexing, similar DNA binding probes could target many different viruses or viral strains on one chip, applied to a single sample. Since each pixel is an independent sensor, even the present chip provides a capacity to multiplex from 1 up to 16,000 probes, and future scalability can readily take this to millions and well beyond (see Scalability below). Methods for constructing such multiplex probe arrays include classic methods, such as fluidic partitioning, or random deposition and combinatorical decoding, such as are used in DNA microarray technology. However, the electronic sensor pixels provide an option of voltage-directed assembly of different probes to specified pixels, by only activating the trapping voltage at pixels of interest as each different probe solution is serially introduced to the array. This would enable precision multiplexing of up to hundreds of probes, due to the speed of active trapping.
Utilizing the full pixel capacity of such chips for massive multiplexing is important for applications such as whole-genome DNA sequencing or whole-proteome profiling, where the anonymous single-molecule targets are captured at each sensor site (DNA target or protein target, respectively) and there undergo many interrogation reactions (polymerase processing, or antibody/aptamer response profiling, respectively) to fully identify or characterize each anonymous DNA or protein target. These extremely high-throughput applications can directly benefit from the multiplexing provided by future chips that scale to millions or even billions of sensors.
Scalability.
The present sensors solve the More-than-Moore scaling problem: the physical extent of the sensors here is defined by the bridge, which is 25 nm long (and which was chosen to be large enough to accommodate all biomolecules of interest as probes). This is already substantially smaller than the minimum metal pitch (MMP, the closest possible metal electrode spacing at the first contact layer used to make contact to transistors) on all existing CMOS fabrication nodes (MPP is ~36 nm on the state-of-the-art [circa 2021] 5 nm CMOS nodes), as well as for all projected nodes spanning the next 10 years (3 nm, 2 nm, 1.4 nm) (75, 76), and thus the size of the molecular element in no way limits the ability to shrink the CMOS pixel circuits (from the current 20 μm pitch reported here, fabricated on a 180 nm CMOS node) in current or foreseeable foundries. Indeed, allowing for reasonable engineering feasibility in existing foundries, future molecular electronic sensor pixels could have a limiting pitch approaching 100 nm, which still allows ample room for the nanoelectrodes contacting vias, and a number of transistors for circuit implementation. This corresponds to a density of 100 million sensors per square millimeter of CMOS chip die. Thus, chips the size of a square millimeter, costing pennies, could provide fantastic sensing capacity. Conversely, a large chip, such as a 10 mm × 10 mm sensor array die, would provide for up to 10 billion sensors.
Rapid Detection.
The single-molecule sensors demonstrated here expose the dynamic nature of single-molecule binding interactions. Just a few seconds of data can survey enough binding events to gather quantitative statistics (eg, Fig. 2B), which can enable extremely rapid measurements and rapid testing (77). In general, near the chemical equilibrium point of the interaction (eg, T m for DNA–DNA binding), the rate of binding nearly equals the rate of unbinding, and numerous events can be observed in a short time, such as the span of seconds. Controlling key reaction variables, such as temperature and target concentration, can be used to adjust the interaction kinetics into such a desirable regime for rapid testing.
Low-Cost Tests.
In many use cases, such as for diagnostics, the CMOS sensor chip would be a single-use disposable. Because of the economy of scale of manufacturing, CMOS chips are extremely low cost when produced at high volume, and therefore support low-cost testing. For example, circa 2021, in the 180 nm CMOS node foundries, medium-to-high production volume, finished commercial CMOS 200 mm wafers cost in the range of $1,000 to $1,400 per wafer, or ~4 cents per square millimeter. (For example, the chip die in the present studies is ~25 mm 2 .) Finer fabrication nodes—such as 65 nm, 22 nm, and 7 nm—are only several-fold more expensive, while enabling orders-of-magnitude higher sensor densities. As shown in Fig. 1B, each square millimeter can contain thousands—and potentially many millions—of sensors (see Scalability above). Thus, molecular electronics chips enable extremely low-cost diagnostic tests.
There have been recent proposals for penny-scale diagnostics relying on low-cost materials, such as PDMS, paper, and ink-jet printed nanoparticles (78⇓–80), yet the extreme economics of CMOS manufacturing allow millions of highly sophisticated sensor circuits to be fabricated on pennies-worth of finished CMOS. The potential of CMOS to provide extremely low-cost diagnostics should not be ignored when paired with general and maximally scalable electronic sensors. In addition to low-cost production, it is important to note that production capacity is unparalleled: the global foundry capacity is estimated to be the equivalent of several hundred million wafers per year, and the industry currently delivers over 1 trillion chips per year. Even for the most extreme imaginable high-volume testing scenarios—such as a future pandemic where such chips are used to test the global population on a near-daily basis—the CMOS chip industry, uniquely, has the required manufacturing capacity. Even for diagnostic concepts based on simple, low-cost materials, in the absence of a manufacturing base it can take decades to reach these production scales.
The instrument needed to run CMOS chip-based assays can be compact, comparable in size to a portable computer or cell phone or USB stick, so that diagnostic tests could be run at the site of use, such as medical point-of-care, or transportation hubs or other public sites, or in homes. For reference, the instrument used for the experimental work reported here (SI Appendix, Fig. S1C) is smaller than a laptop and yet was not optimized for small size. This potential for ultrasmall form factors could support novel environmental sensing methods, such as drone-deployed pathogen sensors actively surveying air or wastewater. Compact, low-cost electronic devices, comparable to a digital thermometer, could also be suitable for home-screening assays of general interest, like health and wellness biomarker panels, early indicators of disease, and at-home diagnostics.
Applications.
These general features of the platform provide support for many applications, with the potential for attractive capabilities and dramatic performance improvements. A few notable examples are discussed here to illustrate this potential.
Molecular electronic microarrays.
The hybridization sensor presented here, deployed in a massively multiplex fashion with many different hybridization oligos represented on one sensor array chip, provides the molecular electronics equivalent of a classic DNA microarray, and could be used for many of the same applications (81⇓–83). However, recast in this framework, it also provides the features of rapid readout and real-time, label-free detection, in an all-electronic format compatible with field deployment on compact devices. Such a next-generation microarray thus confers many benefits. If the oligo probes are taken to be aptamers, this can further provide for diverse targeting, such as protein detection arrays. This illustrates the general principle that mature, classic binding assays, when their molecules are recast as molecular electronic sensors on such a chip platform, inherit many major performance advantages, as well as a long roadmap of further performance improvements.
Drug-target interaction characterization.
The ability to do label-free, time-resolved detection of small-molecule–protein and antibody–antigen interactions enables drug-discovery applications (84). In particular, this may be especially useful for characterizing very weak binding interactions that may represent the earliest stages of drug-candidate selection for poorly druggable targets (85⇓–87). In addition, since the chips are sensitive to single molecules, assays may operate with minimal input materials to support testing of rare compounds. The potential for massive multiplexing on-chip could translate into efficient high-throughput screening of drug candidates, or for molecular evolution programs that rely on screening many mutant protein phenotypes, such as for antibody engineering, developing new CRISPR/Cas genome-editing enzymes, or directed evolution of proteins (68⇓–70, 88).
Diagnostic testing.
The basic sensor types demonstrated here provide a unifying foundation for transferring content from existing molecular diagnostics platforms (77). For example, DNA hybridization is the basis of many forms of nucleic acid detection, such as in qPCR or DNA microarrays, as used in nucleic acid tests for viruses and infectious disease pathogens. Antigen–antibody binding (or aptamer binding) is the basis for immunoassays, as commonly used in lateral flow detection of various antigens, such as pregnancy tests, detection of protein biomarkers, or screening of panels of molecular allergens in the diagnosis of allergy and autoimmune disorders. CRISPR/Cas enzymes have recently been proposed as the basis for new types of diagnostics (64, 66, 67). The sensor examples shown here offer the potential to unify all these disparate diagnostics onto a common chip platform, and provide the benefits of highly multiplex, low-cost tests on a deployment format well-suited to point-of-care testing. Included in this unification is DNA sequencing, which is a diagnostic modality of special importance due to its fundamental role in precision medicine (89).
DNA sequencing.
The binding sensor with a DNA polymerase probe ( Fig. 4 and SI Appendix, Fig. S7) can monitor the activity of the polymerase as it copies a template, with resolution of the individual nucleotide addition events, and discrimination between bases. The ability to monitor a polymerase generally enables “sequencing by synthesis” methods, such as first introduced by Sanger with chain-termination sequencing (90). These methods have dramatically increased in throughput and decreased in cost through the introduction of next-generation massively parallel sequencers (91), and have progressed to single-molecule sequencing platforms (92⇓–94) and CMOS chip-sequencing devices (93⇓–95). The maximal scalability of the present CMOS chip platform provides the potential for further substantial advances in speed and cost. Specifically, for reference, it should be noted that 1 million of the polymerase sensors of Fig. 4 reading bases at accessible Phi29 incorporation rates of 30 to 100 bases per second (73), would generate the quantity of raw sequence data required to read a whole human genome (commonly considered 100 gigabases, or 30x coverage) (91) in under 1 h. CMOS chips with the pixel density of the device shown here (20 μm pitch) (Fig. 1) can have millions of sensors on a large chip, and yet still only cost dollars to produce at high volume. Moreover, the unfettered access to Moore's Law scaling ensures there can also be a long roadmap of continuous improvements in cost and speed. In the present case, the molecular electronic CMOS sensor scaling limits noted above suggest the long-term potential for chips costing tens of pennies, with the sensor capacity to sequence human genomes in tens of seconds. Indeed, it has been noted (96⇓–98) that extending DNA reading capabilities toward such extreme limits is necessary to make storing digital data in DNA an economical option for the future of zettabyte-scale data storage.
Future Developments.
As the scaling potential of the platform is realized, and the diversity of sensors fully developed, the capacity to read vast amounts of molecular interaction data efficiently and economically has the potential to transform applications, such as whole-genome sequencing, whole-proteome profiling, and DNA data storage, and to make diverse diagnostic assays ever more affordable and widely accessible. Through chips such as these, the 50-year-old vision of molecular electronics as the ultimate means of chip miniaturization may be realized, but driven by their value for advancing the power of sensors, rather than processors (13, 77).
Matériaux et méthodes
More detailed materials and methods are in SI Appendix.
Disponibilité des données
All study data are included in main text and SI Appendix.
Remerciements
We thank Sandra Schmid, Eli Yablonovitch, Stan Osher, Stan Nelson, Sue Smalley, and Michael Egholm for helpful discussions and advice, and Mark Reed for providing inspiration and encouragement to pursue Molecular Electronics. This research is supported in part by the Office of the Director of National Intelligence (ODNI), Intelligence Advanced Research Projects Activity (IARPA), through Contract 2019-19081900003 (development of DNA data-storage reading technology). The views and conclusions contained herein are those of the authors and should not be interpreted as necessarily representing the official policies, either expressed or implied, of ODNI, IARPA, or the US government. The US government is authorized to reproduce and distribute reprints for governmental purposes notwithstanding any copyright annotation therein.
Notes de bas de page
Author contributions: CWF, HA, N. Ananthapadmanabhan, TG, DAH, JRS, AGV, S. Jin, JMT, GMC, and BM designed research; CWF, PSP, LA, N. Alagar, N. Ananthapadmanabhan, JB, CC, RD, JF, C. Garcia, C. Gardner, DG, DAH, GH, S. Jain, TJ, RL, ZM, AM, P. Mohan, P. Mola, TN, SO, YO, PP, NP, KP, CR, TR, JRS, PS, JT, AGV, SV, LZ, PWM, and BM performed research; JMT and GMC contributed new reagents/analytic tools; CWF, PSP, SC, KJD, ZG, APH, ZM, P. Mudondo, JM, FO, DP, TS, and BM analyzed data; and CWF, PSP, N. Ananthapadmanabhan, and BM wrote the paper.
Reviewers: JK, University of South Florida College of Arts & Sciences; TL, Seoul National University; and CZ, University of Southern California.
Competing interest statement: All authors having the Roswell affiliation (affiliation “a”) are employed by Roswell Biotechnologies, San Diego, CA 92121.
This article contains supporting information online at https://www.pnas.org/lookup/suppl/doi:10.1073/pnas.2112812119/-/DCSupplemental.
This open access article is distributed under Creative Commons Attribution-NonCommercial-NoDerivatives License 4.0 (CC BY-NC-ND).









